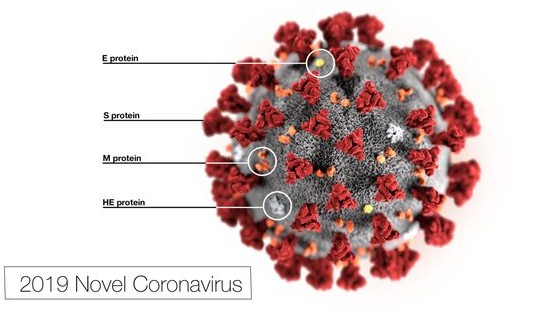
美国国家过敏和传染病研究所疫苗研究中心最近提交的一篇论文对新冠病毒结构进行了研究,并验证了一个结论:新冠病毒 S 蛋白与细胞 ACE2 的亲和力是 SARS 的 10 到 20 倍。
这篇最新论文目前被发布在生物领域预印版论文平台 bioRxiv 上。其中,研究人员通过低温电子显微镜构建了新冠病毒 S 蛋白的预融合构架是三个聚体,发现每个单体上都有细胞受体结合点位。对比新冠与非典的 S 蛋白,研究人员惊讶的发现,新冠病毒和人血管紧张素转化酶 2(ACE2)的结合力是 SARS 病毒的 10-20 倍。这是科学家们首次对于新冠病毒刺突蛋白冷冻电镜结构进行详细观察揭示的秘密。
冠状病毒的刺突糖蛋白(Spike glycoprotein, S glycoprotein)是疫苗、治疗性抗体的研发以及临床诊断的关键靶点。2020 年 2 月 15 日,美国卫生总署 (NIH) 与德克萨斯大学奥斯汀分校 Jason S. McLellan 研究组(此前,McLellan 组在病毒结构方面做了很多重要的工作,研究涉及 MERS 等疾病)进行合作,利用冷冻电镜技术分析了新型冠状病毒表面 S 蛋白的近原子结构。这一研究揭示了新型冠状病毒传染性强的主要原因。
新型冠状病毒大爆发已被宣布为国际关注的突发公共卫生事件。新冠病毒的 S 突刺蛋白也成为疫苗、治疗性抗体研发和诊断的关键靶点。在这篇研究中,研究人员获得了处于预融合构象的新型冠状病毒突刺蛋白三聚体的冷冻电镜(cryo-EM)结构,其分辨率为 3.5Å。三聚体的主要状态为三个受体结合结构域(RBD)之一向上旋转为受体可及构象。生物物理和结构证据表明,较之 SARS 冠状病毒,新型冠状病毒的三聚体更容易与细胞表面的 ACE2 蛋白结合。另外,研究者们也测试了几个已经发表过的 SARS 冠状病毒 RBD 特异性单克隆抗体,发现它们与新冠病毒 S 突刺蛋白没有明显的结合,这表明两种病毒 RBD 之间的抗体交叉反应性可能受到限制。
新型冠状病毒利用高度糖基化的同源三聚体 S 蛋白进入宿主细胞。S 蛋白经历结构变化将病毒融合进入宿主细胞的细胞膜。此过程包括病毒的 S1 亚基结合到宿主细胞受体上,引发三聚体不稳定性的发生,进而造成 S1 亚基脱落 S2 亚基形成高度稳定的融合后结构。为了与宿主细胞受体结合,S1 的受体结合域 (RBD) 经历了类似于铰链的融合构象运动,以暂时隐藏或暴露受体结合的关键位点。这两种状态被称为「下」构象和「上」构象,其中,「下」对应于受体不可结合状态,「上」对应于受体可结合状态,而受体可结合状态被认为较为不稳定。由于 S 蛋白的不可或缺性,它是抗体介入中和的一个脆弱靶点,而预融合 S 结构的特性将为指导疫苗的设计和开发提供原子水平的信息。
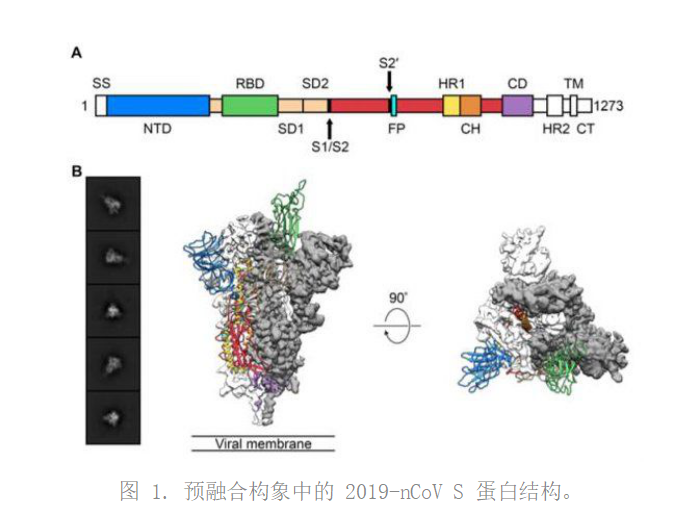
根据新型冠状病毒公布的基因序列,研究者表达了 S 突刺蛋白的胞外域残基(ectodomain residues)1−1208(见图 1A 和补充材料图 1),在基于先前的稳定策略的 C 末端 S2 融合机制中增加了两个稳定脯氨酸突变,该策略被证明对β冠状病毒 S 蛋白高效。
研究者从 FreeStyle 293 细胞中获得了大约 0.5 毫克/升的重组预融合稳定的 S 胞外域,通过亲和层析和凝胶排阻层析进行体外蛋白纯化(补充材料图 1)。使用这种纯化的、完全糖基化的 S 蛋白制备冷冻电镜网格,初步筛选显示高颗粒密度,在孔的边缘几乎没有聚集。
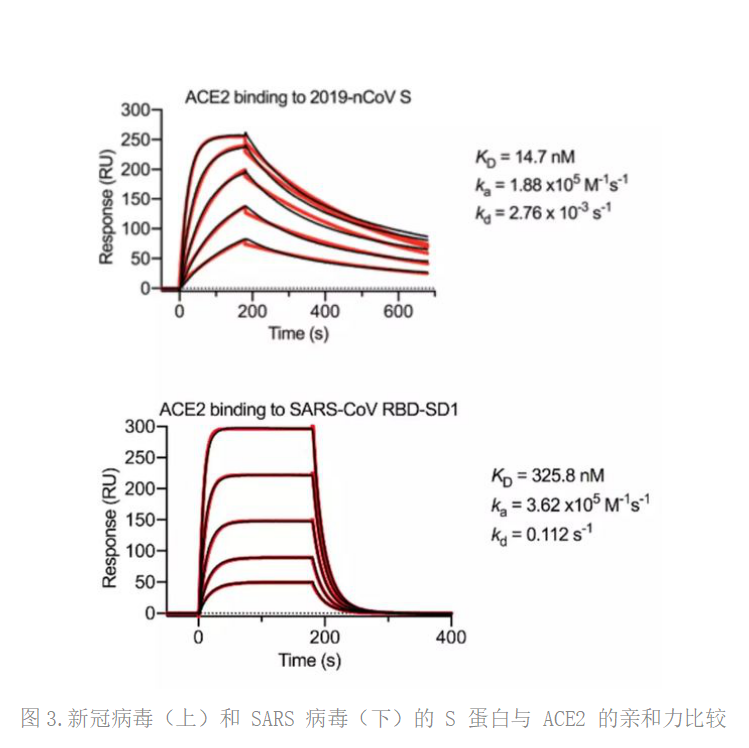
最近的研究表明,新型冠状病毒和 SARS 冠状病毒的 S 蛋白具有相同的功能性宿主细胞血管紧张素转受体(ACE2),这促使我们对于通过表面等离子共振(SPR)的动力学进行量化研究。令人惊讶的是:新冠病毒的 ACE2 胞外域吸引力约为 15nM,相比 SARS 冠状病毒 S 蛋白要高出 10-20 倍。
研究人员还生成了新型冠状病毒 S 蛋白胞外域 ACE2 结合的复合物,并通过负染色对其进行了观察。它与 SARS 冠状病毒 S 蛋白和 ACE2 之间形成的复合物非常相似。新冠病毒对于人类 ACE2 的高亲和力,可能有助于使它在人与人之间的传播变得更加容易,但论文作者认为,还需要进一步的研究来研究这种可能性。
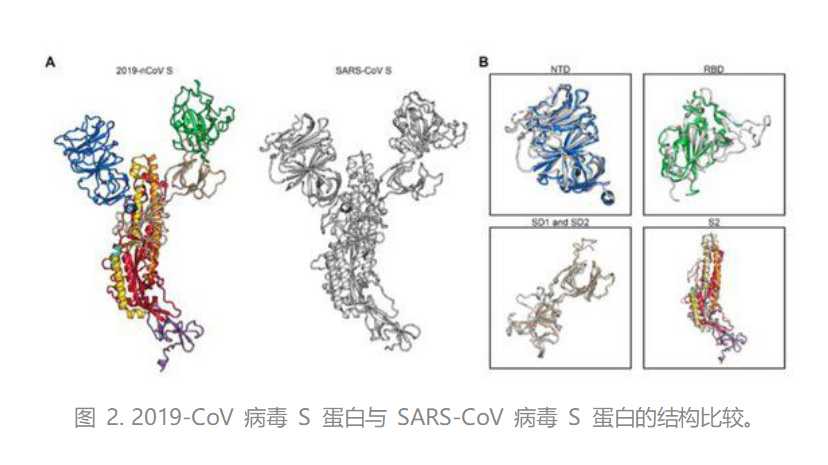
由于新型冠状病毒与 SARS 病毒之间的结构同源性,研究人员希望对已经发表的 SARS 病毒的 RBD 单克隆抗体对新型冠状病毒的 RBD 进行交叉反应性测试。不过,尽管两病毒 RBD 之间结构高度相似,SARS 病毒的 RBD 抗体对于新型冠状病毒并没有明显的结合。SARS RBD 的单克隆抗体对于新型冠状病毒不具有交叉反应,该结果与为新型冠状病毒 S 突刺蛋白作为未来抗体分离与治疗方案的设计提供了重要的参考。
这项研究从近原子水平的分辨率解析了新型冠状病毒 S 蛋白,为进一步精确地疫苗设计以及抗病毒药物的发现提供了重要的结构生物学基础,为后续对抗新冠的疫苗和抗病毒药物研发提供了重要的结构生物学数据支撑。